전체 코드
library(ggplot2)
library(fda)
library(funFEM)
library(plyr)
library(dplyr)
library(reshape)
library(reshape2)
setwd("경로")
mydata <- read.csv("mydata.csv")
test <- mydata %>%
dplyr::select(date, sgg, pop_rate) # 시간, 지역, 인구 추출
datename <- plyr::count(test$date) # 22
datename$id <- rownames(datename)
test <- left_join(test, datename, by=c("date"="x"))
test <- test[c(-4)] # freq 열 살제
test <- dcast(test, date~sgg, value.var = "pop_rate")
test[is.na(test)] <- 0 # na에 0 넣기
test$id <- 1:22 # datename에 따라 수정 필요
times.re <- test[, 1] # date 추출
times <- as.numeric(rownames(test3))
test <- test[, -1]
test <- data.matrix(test)
my_basis <- create.bspline.basis(c(1, 22), breaks=times, norder=5) # datename에 따라 수정 필요
my_par <- fdPar(my_basis, Lfdobj = 2, lambda = 1)
test <- smooth.basis(times, test, my_par)
fdobj <- test$fd
png("test.png",width=4000,height=4000,res=500)
test <- pca.fd(fdobj, nharm = 4)
plot(test$harmonics, lwd = 3)
title(main = "FPCA")
test$varprop
dev.off()
한 줄씩 살펴보면,
library(ggplot2)
library(fda)
library(funFEM)
library(plyr)
library(dplyr)
library(reshape)
library(reshape2)
setwd("경로")
mydata <- read.csv("mydata.csv")필요한 패키지를 불러오고, 경로를 설정한 후 활용할 데이터를 가져오는 과정이다.
test <- mydata %>%
dplyr::select(date, sgg, pop_rate)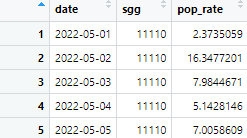
날짜, 지역, 인구 값만 선택하여 test에 입력한다.
datename <- plyr::count(test$date)
datename$id <- rownames(datename)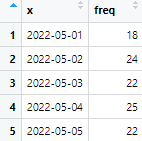
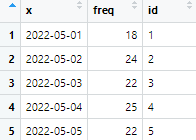
datename에 date 별 데이터의 개수(freq)를 입력한다. 5월 1일에 수집된 지역별 데이터는 18개가 있다. 2022년 5월 1일부터 22일까지 수집된 데이터로, 총 행은 22개다.
datename$id 열을 새로 만들어 datebane의 행 이름(1~22)을 입력한다.
test <- left_join(test, datename, by=c("date"="x"))
test <- test[c(-4)]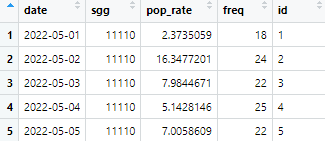
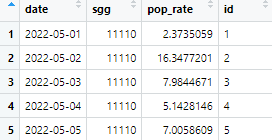
date 값을 조인 키로 활용하여 test에 datename을 조인시킨다.
4번째 열에 해당하는 freq를 삭제한다.
test <- dcast(test, date~sgg, value.var = "pop_rate")
test[is.na(test)] <- 0
test$id <- 1:22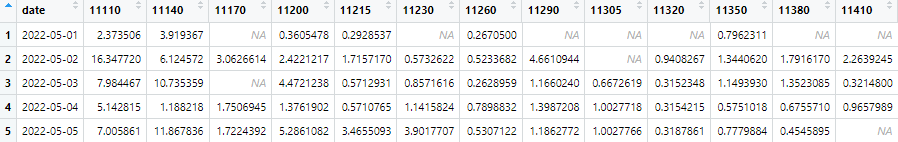
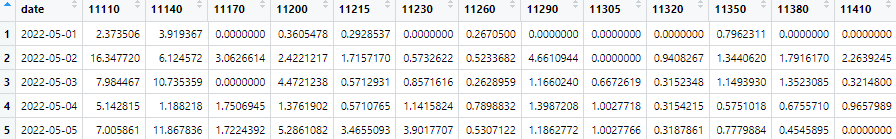
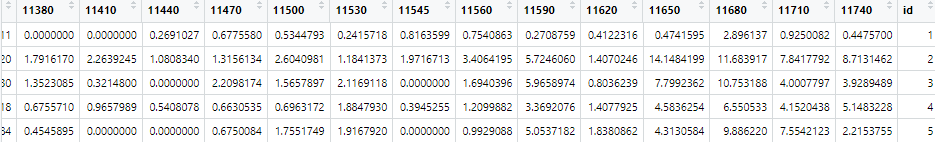
dcast로 교차 테이블을 생성한다.
NA가 있는 곳에 0 값을 넣는다.
id 열을 생성해서 1부터 22까지(datename의 길이와 동일) 값을 입력한다.
times.re <- test[, 1]
times <- as.numeric(rownames(test))
test <- test[, -1]
test <- data.matrix(test)
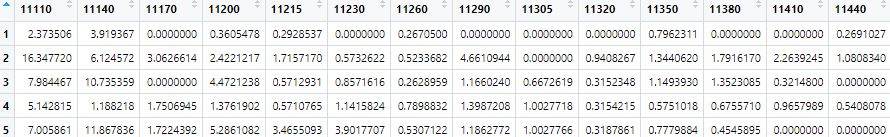

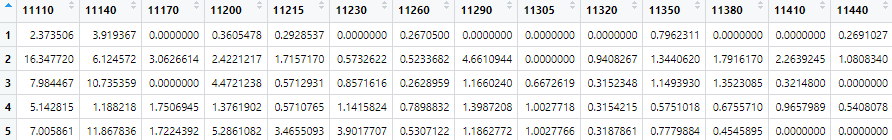
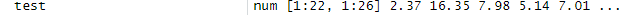
times.re에 날짜 값을 입력한다.
times에 numeric으로 rownames를 입력한다.
test에서 날짜열을 삭제하고, 행렬 데이터로 만든다.
my_basis <- create.bspline.basis(c(1, 22), breaks=times, norder=5)
my_par <- fdPar(my_basis, Lfdobj = 2, lambda = 1)
test <- smooth.basis(times, test, my_par)
fdobj <- test$fd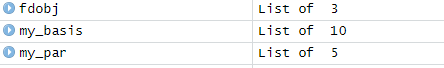

create.bspline.basis 함수를 활용하여 B 스플라인 곡선을 만들어 my_basis에 입력한다. 시간 범위는 1부터 22까지(datename의 길이와 동일)이고, 분할 지점은 times 값, 차수는 5차로 지정한다.
Lfdobj는 평활화 미분, lambda는 평활화 파라미터를 의미한다.
smooth.basis에는 times, test, my_par 인자를 입력한다.
fdobj에는 test의 fd 값을 입력한다.
png("test.png",width=4000,height=4000,res=500)
test <- pca.fd(fdobj, nharm = 4)
plot(test$harmonics, lwd = 3)
title(main = "FPCA")
test$varprop
dev.off()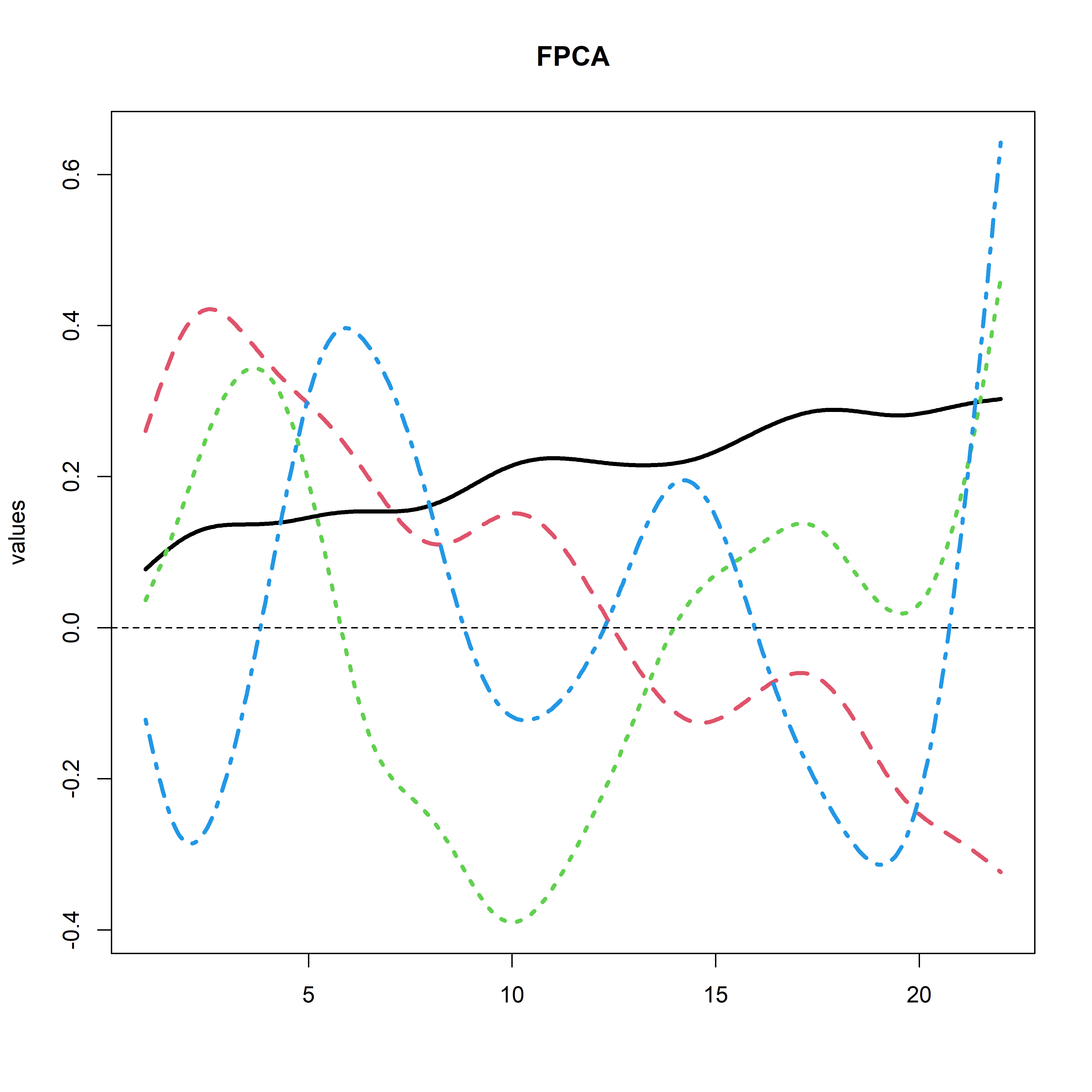
plot을 png으로 저장하는 이 코드들은 한꺼번에 실행해야 한다.
'Data Analysis > R' 카테고리의 다른 글
| [R] rda 파일이 load()로 안열릴 때, readRDS() 활용, rda 파일을 csv로 저장하기 (0) | 2024.05.23 |
|---|
